Figures & data
Tableau 1 Localisation géographique des différentes provenances de Stipa lagascae utilisées dans notre travail. Table 1. Geographic localisation of the different Stipa lagascae ecotypes used in this study.
Fig. 1 Electrophorèse d’ADN génomique de Stipa lagascae sur gel d’agarose 0,8%. (a): ADN extrait par la méthode CTAB 2%; et (b): ADN extrait par la méthode CTAB 3%. Légende: M: marqueur d’ADN (1 kb DNA ladder); 1 à 20: ADN génomique des 20 provenances illustrées dans le tableau I. Fig. 1. Electrophoresis of Stipa genomic DNA on 0.8% agarose gel. (a) DNA extracted by 2% CTAB method and (b): DNA extracted by 3% CTAB method. Legend: M: DNA marker (1Kb DNA ladder); 1-20: Stipa ecotypes genomic DNA illustrated in table I.
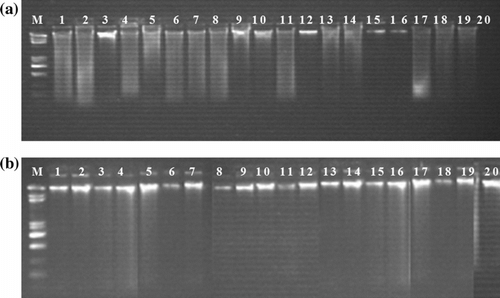
Fig. 2 Optimisation de la quantité d’ADN en PCR. M: Marqueur 100 pb, 1-5 correspondent respectivement à 50; 40; 30; 20 et 10 ng d’ADN. Fig. 2. DNA quantity optimization for PCR. M: 100 pb marker; 1-5 represent 50, 40, 30, 20 and 10 ng of DNA respectively.
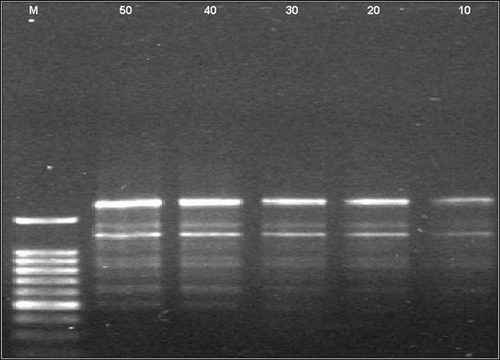
Fig. 3 Profil des produits PCR, sur gel d’agarose 1,5%, en fonction de la concentration en MgCl2. Résultats obtenus avec l’amorce OPJ14 (5’-CACCCGGATG-3’) chez S. lagascae. M: marquer de taille (1Kb); 1: 0,5 mM; 2: 1 mM; 3: 1,5 mM; 4: 2 mM; 5: 2,5 mM; 6: 3 mM et 7: 3,5 mM. Fig. 3. Profile of PCR products on 1.5% agarose gel, depending on the concentration of MgCl2. Results obtained with primer OPJ14 (5’-CACCCGGATG-3’) in S. lagascae. M: 1Kb marker, 1: 0.5 mM, 2: 1 mM, 3: 1.5 mM, 4: 2 mM, 5: 2.5 mM, 6: mM and 7: 3.5 mM.
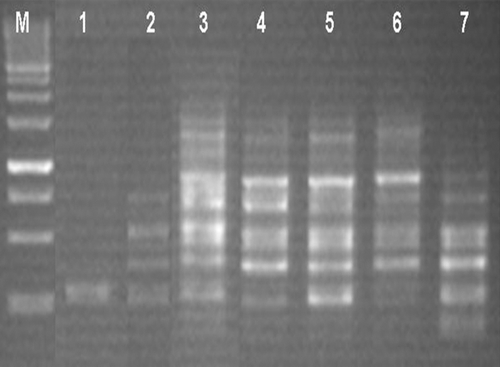
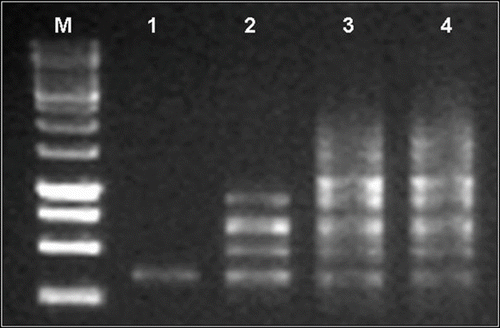
Fig. 4 Profil RAPD de quatre concentrations de l’amorce sur gel d’agarose 1,5%: résultats obtenus avec l’amorce OPJ14 (5’- CACCCGGATG -3’) chez S. lagascae. M: marquer de taille (1Kb); 1: 10 pM; 2: 20 pM; 3: 30 pM et 4: 40 pM. Fig. 4. RAPD profile obtained from different concentrations of primer OPJ 14 (5’- CACCCGGATG -3’) for S. lagascae. M: 1 Kb marker, 1: 10 pM; 2: 20 pM; 3: 30 pM and 4: 40 pM.
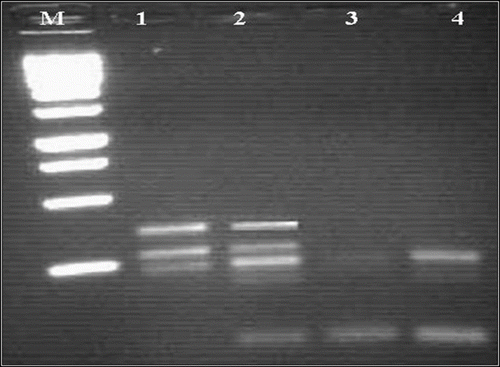
Fig. 5 Profil des produits PCR, sur gel d’agarose 1,5%, en fonction de la concentration en Taq Polymérase. Résultats obtenus avec l’amorce OPJ14 (5’- CACCCGGATG -3’) chez S. lagascae. M: marquer moléculaire (1Kb); 1: 0,5 U; 2: 1 U; 3: 1,5 U et 4: 2 U. Fig. 5. PCR products profile on 1.5% agarose gel of different concentration of Taq polymerase. Results obtained with primer OPJ14 (5’- CACCCGGATG -3’) for S. lagascae. M: 1Kb marker, 1: 0.5 U; 2: 1U, 3: 1,5 and 4 U: 2 U.