Résumé
L’isolement de l’ADN génomique intact est une étape essentielle dans différentes applications de la biologie moléculaire. Dans de nombreuses plantes, la qualité de l’ADN extrait n’est pas appréciée en raison de métabolites secondaires qui interfèrent avec les procédures d’isolement de l’ADN et les réactions PCR. Dans cette étude, nous avons développé une méthode efficace et fiable pour isoler l’ADN génomique de Stipa lagascae Roem. & Schult. dépourvu de polysaccharides et de polyphénols. Les résultats ont montré que 3% de CTAB (50 mM Tris (pH 8), 3 M de NaCl, 1% PEG 4000, 3% PVPP, 0,5 M glucose, 50 mM EDTA et 0,2% ß-mercaptoéthanol) a généré de l’ADN de haute qualité confirmée par des analyses spectrophotométriques (un rapport de 1,81 pour DO260/DO280). De même, la quantité de 40 à 50 ng d’ADN semblait suffisante pour les réactions PCR. En outre, 20 pmoles/25μl d’amorce; 2,5 mM de chlorure de magnésium et 1U de Taq polymérase sont les concentrations les plus fiables pour générer des profils clairs et reproductibles afin d’évaluer la diversité génétique parmi les différentes accessions de Stipa lagascae Roem. & Schult.
Abstract
The isolation of intact, high-molecular-mass genomic DNA is essential for many molecular biology. In many plants, the quality of the extracted DNA is not appreciated because of metabolites that interfere with DNA isolation procedures and subsequent applications. In this study we developed an efficient and reliable method for isolating Stipa lagascae Roem. & Schult. DNA free from polysaccharides and polyphenols. Results showed that CTAB 3% (50 mM Tris (pH 8); 3 M NaCl; 1% PEG 4000; 3% PVPP; 0.5 M glucose; 50 mM EDTA and 0.2% ß-mercaptoéthanol) generated high quality DNA confirmed by spectrophotometric analyses (DO260/DO280 ratio of 1.81). 40 to 50 ng of DNA seemed to be adequate quantity for PCR reactions. Furthermore, 20pmoles/25μl of primer; 2.5 mM of magnesium chloride and 1U of Taq polymerase are the most reliable concentrations to have clear and reproducible profiles to assess the genetic diversity among different accessions of Stipa lagascae Roem. & Schult.
Introduction
Les marqueurs morphologiques contrôlés par un seul locus peuvent être considérés comme des marqueurs génétiques si leurs expressions sont reproductibles sous des conditions environnementales différentes (Staub et al., Citation1996). Malgré le fait que les marqueurs morphologiques codominants ont été utilisés comme des prédicateurs de la réponse génétique à la sélection, ils peuvent être influencés par des facteurs environnementaux (facteurs climatiques, édaphiques et biotiques) et génétiques (épistasie). Les marqueurs biochimiques ‘allozymes’ sont spécifiques aux tissus végétaux et les résultats sont souvent difficiles à interpréter lorsque différentes formes isoenzymatiques présentent la même mobilité électrophorétique (Murphy et al., Citation1996). Quoique relativement peu coûteuse, l’électrophorèse enzymatique nécessite habituellement des quantités relativement importantes de tissus, rendant les études allozymiques moins faciles pour les taxons rares. Contrairement aux marqueurs traditionnels (morphologiques et biochimiques), les marqueurs moléculaires ne sont pas influencés par les fluctuations de l’environnement et sont indépendants de l’organe analysé et du stade de développement de la plante (Najimi et al., Citation2003). Par ailleurs, ces marqueurs sont utilisés pour décrire la variabilité génétique et sa répartition au sein de populations et d’espèces, comme ils servent à préciser les mécanismes évolutifs des populations qui rendent compte de cette description (Santoni et al., Citation2000). Ces marqueurs moléculaires deviennent aujourd’hui un outil essentiel d’amélioration des plantes et ouvrent des nouvelles perspectives pour le sélectionneur (Najimi et al., Citation2003). En fait, l’étude de la diversité génétique des lignées au niveau moléculaire permet de comprendre les différences et les similitudes qui existent entre ces lignées au niveau de leurs ADN et de décrire la structuration de leur variabilité génétique. Ceci peut avoir plusieurs applications: une meilleure gestion des ressources génétiques sur la base d’une évaluation moléculaire complémentaire de l’évaluation agronomique, le choix des lignées ou les accessions génétiquement éloignées et complémentaires pour la réalisation des croisements de départ dans les programmes de sélection et enfin situer, à tout moment, les variétés nouvellement introduites par rapport à celles déjà utilisées pour une meilleure protection des droits d’obtention de nouvelles variétés (distinction variétale) (Rafalsky et al., 1991; Moreau et al., Citation2001).
Parmi les marqueurs moléculaires, la RAPD est une technique efficace pour l’étude de la diversité génétique inter et intra population (Williams et al., Citation1990). Elle a l’avantage d’être rapide et facile, nécessitant peu de matériel végétal, et peut être utilisée à moindre coût pour détecter le polymorphisme génétique des organismes (Williams et al., Citation1990). Cette méthode moléculaire, relativement peu coûteuse, a été largement utilisée dans la recherche fondamentale et appliquée dans de nombreuses familles de plantes différentes (Bussell, Citation1999; Chen et al., Citation2005; Ge et al., Citation1998; Gustafson et al., Citation2002, Citation2004).
Stipa lagascae Roem. & Schult. est une Poaceae pérenne, autochtone, à répartition géographique circum-méditerranéenne (Scholz, Citation1991). Ce taxon est réputé résistant au pâturage, et surtout apte à la repousse même après de très faibles précipitations (Chaieb et al., Citation1992). S’agissant d’une espèce hautement appétée et très recherchée par les ruminants au niveau des steppes présahariennes (précipitations moyennes annuelles de 100-200 mm) (Le Houérou & Ionesco, Citation1973), cette espèce est jusqu’ici considérée prometteuse pour les opérations de re-semis et de restauration des écosystèmes (Aronson et al., Citation1993). Elle est très connue pour son polymorphisme phénotypique en combinaison avec la cléistogamie (Visser et al., Citation2001; Ouled Belgacem et al., Citation2006). Avant d’entamer l’étude de la diversité génétique de Stipa lagascae moyennant les marqueurs RAPD, l’optimisation du protocole d’extraction de l’ADN et l’optimisation de la PCR doivent être réalisées pour réduire le coût des analyses.
Matériel et méthodes
Matériel végétal
Le matériel végétal utilisé dans cette étude est représenté par 20 provenances de Stipa (Stipa lagascae Roem. & Schult.) dont les semences ont été collectées à partir d’une collection de base, installée dans la parcelle expérimentale située au siège de l’Institut des Régions Arides- Médenine, à la suite d’une prospection effectuée en 2001. Cette sélection a été faite en se basant sur la vigueur végétative et reproductive de ces provenances (Visser et al., Citation2001). Les provenances étudiées représentent plusieurs localités du centre et du sud de la Tunisie (Tableau ).
Tableau 1 Localisation géographique des différentes provenances de Stipa lagascae utilisées dans notre travail. Table 1. Geographic localisation of the different Stipa lagascae ecotypes used in this study.
Méthodes
Extraction de l’ADN
L’extraction et la purification de l’ADN total à partir d’une plante constituent la première étape importante pour la caractérisation des végétaux. Dans notre étude, l’extraction de l’ADN total a été faite à partir de 200 mg de matière fraîche d’échantillons de jeunes feuilles, conservées à -80 °C, en présence de l’azote liquide et 3 ml du tampon CTAB. Nous avons essayé d’optimiser la méthode d’extraction de l’ADN en essayant deux concentrations en CTAB (2 et 3%). Le premier tampon d’extraction est composé de 2% CTAB, 200 mM Tris (pH 8), 1,5 M NaCl, 20 mM EDTA et de 0,2% de ß-mercaptoéthanol. Le deuxième est composé de 3% CTAB, 250 mM Tris (pH 8), 3 M NaCl, 1% de PEG 4000, 3% PVPP, 0,5 M de glucose, 50 mM EDTA et de 0,2% de ß-mercaptoéthanol. L’ADN a été précipité en présence de l’isopropanol pendant une nuit à -20 °C et il a été purifié par un traitement à la RNAse et à la protéinase K.
Quantification de l’ADN
La quantification de l’ADN ainsi que la vérification de sa qualité ont été faites par observation sur gel d’agarose et coloration au bromure d’éthidium, par comparaison à la fluorescence d’un autre ADN de concentration connue, et par spectrophotométrie à une longueur d’onde de 260 puis à 280 nm pour en déduire le rapports DO260/DO280 (rapport estimant la pureté de l’ADN).
Optimisation de la réaction PCR
Qualité et quantité d’ADN matriciel. L’usage qui sera fait de l’ADN (stockage ou non) et la nature du prélèvement conditionnent souvent le protocole de préparation qui sera utilisé. Néanmoins, le pré-requis à toute amplification est que l’ADN utilisé comme matrice soit débarrassé d’inhibiteurs potentiels de la PCR. La Taq polymérase, enzyme thermorésistante, présente en effet des sensibilités variables à certains agents chimiques et biologiques (éthanol, phénol, EDTA, protéines, ARN, etc.) (Scott & Playford, Citation1996; Sablok et al., Citation2009). Ces inhibiteurs proviennent soit de l’échantillon, soit des réactifs utilisés dans les protocoles d’extraction et de conservation.
La quantité utilisée d’ADN peut aussi influer sur le résultat d’amplification, une quantité insuffisante d’ADN ne permet pas d’obtenir d’amplification, alors qu’une quantité trop importante d’ADN peut inhiber la réaction à cause de l’accumulation de contaminants (Vroh Bi et al., Citation1997). Nous avons donc testé différentes quantités d’ADN (50, 40, 30, 20 et 10 ng).
Qualité et quantité des amorces. La réaction PCR nécessite l’hybridation d’amorces spécifiques sur la séquence que l’on souhaite amplifier. Cette hybridation spécifique est conditionnée notamment par la qualité et la quantité des amorces utilisées. Une gamme de concentration (10, 20, 30, 40, 50 et 60 μM) a été étudiée pour choisir la concentration la plus adéquate.
Quantité de MgCl2 . L’ion Mg2+ est un cofacteur essentiel de la Taq polymérase. Ce cation bivalent interagit également avec les charges négatives de la chaîne d’ADN, limitant ainsi les forces de répulsion entre brins d’ADN et favorisant donc la stabilité de l’hybridation. Plus sa concentration est importante, plus l’hybridation est facilitée, que celle-ci soit spécifique ou non. Une trop forte concentration peut alors conduire à une augmentation des signaux non spécifiques. Pour optimiser la réaction PCR nous avons étudié une gamme de concentration de Mg2+ (0,5; 1; 1,5; 2; 2,5; 3 et 3,5 mM).
Quantité de Taq polymérase
Pour ce qui est des quantités d’enzymes à utiliser, nous avons testé 0,5U; 1 U; 1,5 U et 2 U. Afin de réduire l’erreur expérimentale, chaque échantillon a été dupliqué en deux.
La réaction PCR
La réaction d’amplification a été réalisée dans un volume de 25 μl contenant 40 ng d’ADN génomique, 0,2 mM dNTPs, 0,25 μM d’amorces, 1 unité de Taq polymerase (GoTaq, Promega) et son tampon concentré une fois, 2,5 mM MgCl2 et l’eau ultra pure. Les cycles suivants furent réalisés: 94 °C/3 min, 40 X (94 °C/1 min, 36 °C/0,45 min, 72 °C/1 min.), 72 °C/7 min. Pour chaque réaction PCR un contrôle négatif (sans ADN) est toujours intégré.
Résultats et discussions
Quantification de l’ADN
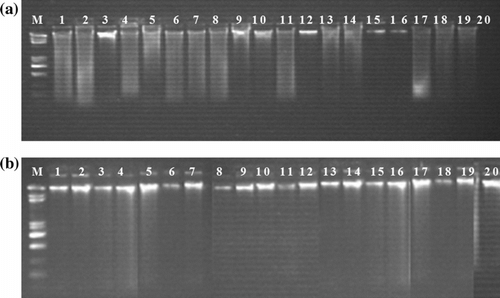
L’ADN extrait des différentes provenances a été quantifié par spectrophotométrie et par électrophorèse sur gel d’agarose 0,8% sous un voltage constant de 80 v. L’extraction par le CTAB concentré (3%) a donné une meilleure qualité de l’ADN que celle faite par le CTAB (2%). L’utilisation du CTAB 2% a donné un rendement important en ADN mais avec présence de polysacharides, ce qui a engendré un rapport faible de la DO260/DO280 estimant le degré d’impureté de l’ADN extrait. Cependant, le tampon CTAB 3% concentré en sel contenant le PEG et le PVPP a donné un ADN pur, facilement amplifiable par PCR et adéquat pour les différentes techniques moléculaires avec un rendement important (Fig. ).
Fig. 1 Electrophorèse d’ADN génomique de Stipa lagascae sur gel d’agarose 0,8%. (a): ADN extrait par la méthode CTAB 2%; et (b): ADN extrait par la méthode CTAB 3%. Légende: M: marqueur d’ADN (1 kb DNA ladder); 1 à 20: ADN génomique des 20 provenances illustrées dans le tableau I. Fig. 1. Electrophoresis of Stipa genomic DNA on 0.8% agarose gel. (a) DNA extracted by 2% CTAB method and (b): DNA extracted by 3% CTAB method. Legend: M: DNA marker (1Kb DNA ladder); 1-20: Stipa ecotypes genomic DNA illustrated in table I.
Optimisation de la réaction PCR
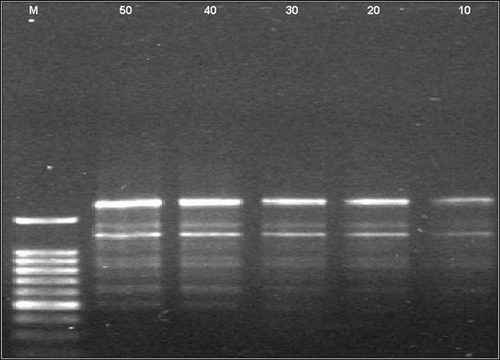
Effets des différentes concentrations d’ADN
La concentration en ADN dans le milieu réactionnel est un facteur important qui peut influencer la réaction PCR. D’après les résultats présentés dans la figure , les quantités 50 et 40 ng.μl-1 ont donné de meilleurs résultats que ceux obtenus par les quantités moindres.
Effets des différentes concentrations en MgCl2
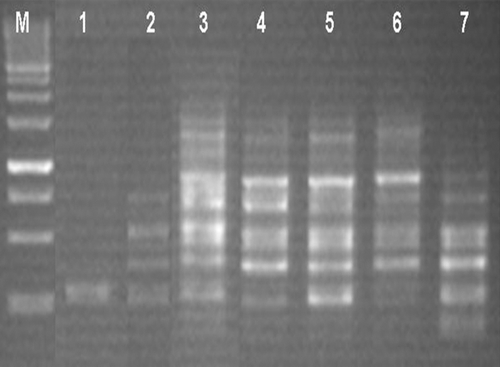
La figure montre la différence d’amplification observée à quatre concentrations différentes de MgCl2. La réaction a été réalisée avec l’amorce OPJ-14 (CACCCGGATG avec Tm = 34) sur l’ADN de la provenance 12. La réduction drastique du nombre de bandes et leur intensité confirment l’importance du rôle du MgCl2 dans la réaction PCR et l’intérêt d’en optimiser leur concentration. Le chlorure de magnésium est un élément essentiel dans la réaction PCR. Les ions Mg2+ forment un complexe soluble avec les dNTPs, indispensable à l’incorporation de ces derniers dans la chaîne d’ADN en croissance; ces ions stimulent l’activité de l’ADN polymérase (Newton & Graham, Citation1994). De plus, MgCl2 affecte d’autres paramètres comme l’hybridation des amorces et la dissociation des brins d’ADN et des produits d’amplification. Dans nos expériences, 0,5; 1; 1,5; 2; 2,5; 3 et 3,5 mM de MgCl2 ont été testés. Les concentrations 2 et 2,5 ont donné les résultats les plus fiables. La concentration 2,5 mM a été utilisée pour la suite de nos travaux.
Fig. 3 Profil des produits PCR, sur gel d’agarose 1,5%, en fonction de la concentration en MgCl2. Résultats obtenus avec l’amorce OPJ14 (5’-CACCCGGATG-3’) chez S. lagascae. M: marquer de taille (1Kb); 1: 0,5 mM; 2: 1 mM; 3: 1,5 mM; 4: 2 mM; 5: 2,5 mM; 6: 3 mM et 7: 3,5 mM. Fig. 3. Profile of PCR products on 1.5% agarose gel, depending on the concentration of MgCl2. Results obtained with primer OPJ14 (5’-CACCCGGATG-3’) in S. lagascae. M: 1Kb marker, 1: 0.5 mM, 2: 1 mM, 3: 1.5 mM, 4: 2 mM, 5: 2.5 mM, 6: mM and 7: 3.5 mM.
Effets des différentes concentrations de l’amorce
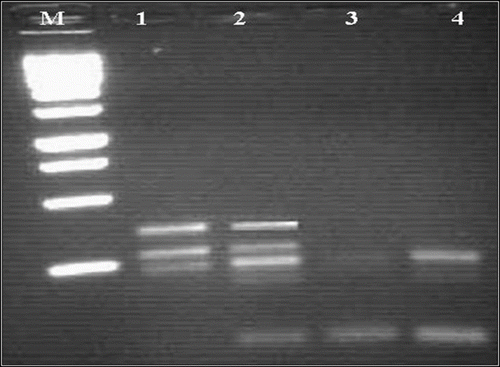
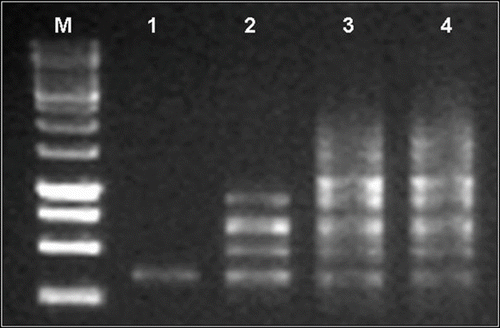
Les résultats illustrés dans la figure montrent que la meilleure amplification est obtenue avec 20 pmoles/25μl. Au-delà, les ‘smears’ apparaissent avec perte des grands fragments.
Fig. 4 Profil RAPD de quatre concentrations de l’amorce sur gel d’agarose 1,5%: résultats obtenus avec l’amorce OPJ14 (5’- CACCCGGATG -3’) chez S. lagascae. M: marquer de taille (1Kb); 1: 10 pM; 2: 20 pM; 3: 30 pM et 4: 40 pM. Fig. 4. RAPD profile obtained from different concentrations of primer OPJ 14 (5’- CACCCGGATG -3’) for S. lagascae. M: 1 Kb marker, 1: 10 pM; 2: 20 pM; 3: 30 pM and 4: 40 pM.
Effets des différentes concentrations en Taq polymérase
D’après les résultats présentés dans la figure , la quantité inférieure à une unité entraîne la perte de plusieurs fragments. Entre 1,5 et 2 U, les amplifications semblent être constantes.
Fig. 5 Profil des produits PCR, sur gel d’agarose 1,5%, en fonction de la concentration en Taq Polymérase. Résultats obtenus avec l’amorce OPJ14 (5’- CACCCGGATG -3’) chez S. lagascae. M: marquer moléculaire (1Kb); 1: 0,5 U; 2: 1 U; 3: 1,5 U et 4: 2 U. Fig. 5. PCR products profile on 1.5% agarose gel of different concentration of Taq polymerase. Results obtained with primer OPJ14 (5’- CACCCGGATG -3’) for S. lagascae. M: 1Kb marker, 1: 0.5 U; 2: 1U, 3: 1,5 and 4 U: 2 U.
La technique RAPD est un outil très utile d’assistance en amélioration des plantes grâce à sa rapidité et à sa simplicité par rapport aux autres types de marqueurs moléculaires. Les conditions optimales pour la réalisation de la PCR varient sensiblement en fonction du matériel végétal utilisé. L’optimisation des différents paramètres intervenant dans cette technique constitue un préalable indispensable à toute utilisation de ce type de marqueurs dans la conservation du patrimoine génétique et dans les programmes futurs d’amélioration. Ce travail préliminaire doit aboutir à un rendement optimal en termes d’amplification de l’ADN et garantir une haute reproductibilité des résultats. Cependant, il est souhaitable de partir des conditions proposées par Williams et al. (Citation1990) et de tenir compte des conditions proposées par les fournisseurs des différents constituants de la réaction pour faire ensuite l’optimisation. Dans notre étude, les concentrations adéquates d’ADN pour une meilleure amplification sont de 30 à 100 ng. Zhao et al. (Citation2006a) et Wang et al. (Citation2006a) ont utilisé 30 ng d’ADN de Stipa pour la méthode RAPD. Lors de nos expérimentations, les faibles concentrations en MgCl2 (Fig. : pistes 1, 2 et 3) et les fortes concentrations (Fig. : pistes 6 et 7) ont généré une perte de bandes. La concentration 2,5 mM semble être la plus adéquate. Cette concentration a été utilisée par plusieurs chercheurs travaillant sur Stipa (Turuspekov et al., Citation2002; Zhao et al., Citation2008). Les amorces constituent un facteur essentiel dans la réaction PCR. Une concentration de 20 pM/25μl donne des résultats fiables avec des bandes nettes. Ces résultats corroborent avec ceux trouvés par Vroh Bi et al. (Citation1997) travaillant sur le cotonnier.
Pour les différentes concentrations de Taq, les résultats montrés par différents chercheurs (Vroh Bi et al., Citation1996; Boussaha & Ben Naceur, Citation2009) suggèrent que les Taq polymérases provenant de différents fournisseurs peuvent avoir des comportements différents. Plusieurs études faites sur Stipa ont montré que 1U de Taq est la concentration adéquate (Turuspekov et al., Citation2002; Zhao et al., Citation2006b; Zhao et al., Citation2008; Wang et al., Citation2006a, 2006b). Pour des raisons d’économie, nous avons choisi 1U pour la suite de notre étude.
L’optimisation du protocole d’extraction de l’ADN et des différents paramètres de la PCR constitue une étape très importante avant d’entamer l’étude de la diversité génétique de Stipa lagascae.
Conclusion
Cette étude nous a permis de mettre au point une technique d’extraction d’un ADN de très bonne qualité à partir des jeunes feuilles de Stipa lagascae. L’optimisation des conditions de la réaction PCR pour l’amplification des RAPD nous a permis d’avoir des profils clairs et reproductibles afin d’évaluer la diversité génétique entre différentes accessions localisées à l’IRA.
Références
- Aronson , J. , Floret , C. , Le Floc’h , E. , Ovalle , C. and Pontanier , R. 1993 . Restoration and rehabilitation of degraded ecosystems in arid and semi-arid lands. II Case studies in Southern Tunisia, Central Chile and Northern Cameroon . Restor Ecol. , 1 : 168 – 187 .
- Boussaha , A. and Ben Naceur , M. 2009 . Optimisation de la technique RAPD et étude de la diversité génétique de quelques accessions d’Oxalis corniculata L . Revue des régions arides. , 22 : 73 – 85 .
- Bussell , J.D. 1999 . The distribution of random amplified polymorphic DNA (RAPD) diversity among populations of Isotoma petraea (Lobeliaceae) . Mol Ecol. , 8 : 775 – 789 .
- Chaieb , M. , Floret , C. , Le Floch , E. and Pontanier , R. 1992 . Life history strategies and water resource allocation in five pasture species of the Tunisian arid zone . Arid Soil Res Rehab. , 6 : 1 – 10 .
- Chen , S.L. , Xia , T. , Chen , S.Y. and Zhou , Y.J. 2005 . RAPD profiling in detecting genetic variation in endemic Coelonema (Brassicaceae) of Qinghai-Tibet Plateau of China . Biochem Genet. , 43 : 189 – 201 .
- Ge , S. , Hong , D.Y. , Wang , H.Q. , Liu , Z.Y. and Zhang , C.M. 1998 . Population genetic structure and conservation of an endangered conifer, Cathaya argyrophylla (Pinaceae) . Int J Plant Sci. , 159 : 351 – 357 .
- Gustafson , D.J. , Gibson , D.J. and Nickrent , D.L. 2002 . Genetic diversity and competitive abilities of Dalea purpurea (Fabaceae) from remnant and restored grasslands . Int J Plant Sci. , 163 : 979 – 990 .
- Gustafson , D.J. , Gibson , D.J. and Nickrent , D.L. 2004 . Conservation genetics of two co-dominant grass species in an endangered grassland ecosystem . J Appl Ecol. , 41 : 389 – 397 .
- Houérou H.N. Le & T. Ionesco, 1973. Appétabilité des espèces végétales de la Tunisie steppique. Doc. travail ronéoté, 68 p.
- Moreau , L. , Charcosset , A. and Gallais , A. 2001 . Efficiency of marker-assisted selection compared with conventional selection . Corps Gras Lipides. , 8 ( 5 ) : 496 – 501 .
- Murphy , R.W. , Sites , J.W. , Buth , D.G. and Haufler , C.H. 1996 . “ Proteins: isozyme electrophoresis ” . In Sinauer Associates , 2nd edn , Edited by: Systematics , Molecular , Hillis , D.M. , Moritz , C. and Mable , B.K. 51 – 20 . MA : Sunderland .
- Najimi , B. , El Jaafari , S. , Jlibène , M. and Jacquemin , J.M. 2003 . Applications des marqueurs moléculaires dans l’amélioration du blé tendre pour la résistance aux maladies et aux insectes . Biotechnol. Agron. Soc. Environ. , 7 ( 1 ) : 17 – 5 .
- Newton CR. & A. Graham, 1994. ‘‘PCR’’, pp. 1–161. Bios scientific Publishers, Oxford, UK.
- Ouled Belgacem , A. , Neffati , M. , Papanastasis , V.P. and Chaieb , M. 2006 . Effects of seed age and seedling depth on growth of Stipa lagascae R. and Sch. seedlings . J Arid Environ. , 65 : 682 – 687 .
- Rafalski , J.A. , Tingey , S.V. and Williams , J.G.K. 1991 . RAPD markers A new technology for genetic mapping and plant breeding . AgBiotech News and Information. , 3 : 645 – 648 .
- Sablok , G. , Gahlot , P. , Gupta , A.K. , Pareek , K. and Shekhawat , N.S. 2009 . Extraction of PCR-usable DNA from trees adapted to arid environment . Plant Omics Journal. , 2 ( 3 ) : 103 – 109 .
- Santoni , S. , Faivre-Rampant , P. , Prado , E. and Prat , D. 2000 . Marqueurs moléculaires pour l’analyse des ressources génétiques et l’amélioration des plantes . Cahiers Agricultures. , 9 ( 4 ) : 311 – 327 .
- Scholz , H. 1991 . Stipa tunetana, eine neue Art aus Tunesien, und das St. Lagascae Aggregat (Gramineae) . Willdenowia. , 26 : 225 – 228 .
- Scott , K.D. and Playford , J. 1996 . DNA extraction technique for PCR in rainforest plant species . Biotechniques. , 20 : 974 – 978 .
- Staub , J.E. , Serquen , F.C. and Gupta , M. 1996 . Genetic markers, map construction and their application in plant breeding . Hortic Sci. , 31 : 729 – 741 .
- Turuspekov , Y. , Adams , R.P. and Kearney , C.M. 2002 . Genetic diversity in three perennial grasses from the Semipalatinsk nuclear testing region of Kazakhstan after long-term radiation exposure . Biochem Syst Ecol. , 30 : 809 – 817 .
- Visser , M. , Morand , F. and Mahdhi , H. 2001 . Fauche de Stipa lagascae et réhabilitation des terres privées en Tunisie présaharienne . Cah Agric. , 11 : 377 – 383 .
- Vroh Bi , I. , Harvengt , L. , Chandellier , A. and Mergeai , G. 1996 . Improved RAPD amplification of recalcitrant plant DNA by the use of activated charcoal during DNA extraction . Plant Breed. , 115 : 205 – 206 .
- Vroh Bi , I. , du Jardin , P. , Mergeai , G. and Baudoin , J.P. 1997 . Optimisation et application de la RAPD (Random Amplified Polymorphic DNA) dans un programme de sélection récurrente chez le cotonnier (Gossypium spp.) . Biotechnol Agron Soc Environ. , 2 : 142 – 150 .
- Wang J.L., N.X. Zhao & Y.B. Gao, 2006a. RAPD Analysis of Genetic Diversity and Population Genetic Structure of Stipa krylovii Reshov. in Inner Mongolia Steppe. Russ J Genet+. 42, 468–475.
- Wang , J.L. , Gao , Y.B. and Zhao , N.X. 2006b . Morphological and RAPD analysis of the dominant species Stipa krylovii Roshev . In Inner Mongolia steppe. Bot Stud. , 47 : 23 – 35 .
- Williams , J.G. , Kubelik , A.R. , Livak , K.J. , Rafalski , J.A. and Tingey , S.V. 1990 . DNA polymorphisms amplified by arbitrary primers are useful as genetic markers . Nucleic Acids Res. , 18 : 6531 – 6535 .
- Zhao , N.X. , Gao , Y.B. , Wang , J.L. , Ren , A.Z. and Hua , X. 2006a . RAPD diversity of Stipa grandis populations and its relationship with some ecological factors . Acta Ecol Sin. , 26 ( 5 ) : 1312 – 1319 .
- Zhao , N.X. , Gao , Y.B. , Wang , J.L. and Ren , A.Z. 2006b . Genetic Diversity and Population Differentiation of the Dominant Species Stipa krylovii in the Inner Mongolia Steppe . Biochem Genet. , 44 : 513 – 526 .
- Zhao , N.X. , Gao , Y.B. , Wang , J.L. and Ren , A.Z. 2008 . Population structure and genetic diversity of Stipa grandis P. Smirn, a dominant species in the typical steppe of northern China . Biochem Syst Ecol. , 36 : 1 – 10 .