Figures & data
Table 1. Sequences of primers for real-time PCR in the study.
Tabla 1. Secuencias en tiempo real de cebadores para la PCR en este estudio.
Table 2. The molecular weight and molecular weight of OKGM and U-OKGM.
Tabla 2. Peso molecular de OKGM y U-OKGM.
Figure 1. MW measurement of OKGM and U-OKGM by GPC-MALLS with the laser spectrum.
Figura 1. Medición MW de OKGM y U-OKGM por medio de la GPC-MALLS con la frecuencia espectral de láser.
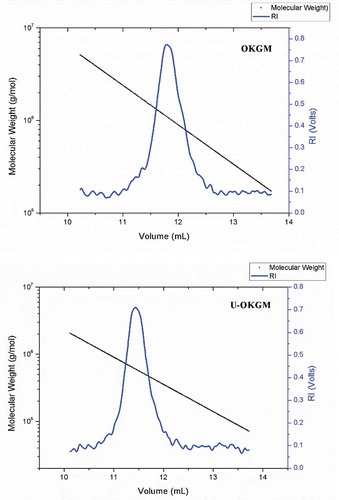
Figure 2. Scanning electron micrograph of OKGM and U-OKGM (×100 times).
Figura 2. Micrografía electrónica de barrido de OKGM y U-OKGM (×100 veces).

Figure 3. FT-IR spectrum of OKGM and U-OKGM. OKGM: oxidized konjac glucomannan; U-OKGM: ultrasound degradated-OKGM.
Figura 3. Espectro FT-IR de OKGM y U-OKGM. OKGM: glucomanano de konjac oxidado; U-OKGM: OKGM degradado por ultrasonido.
Figure 5. Effect of U-OKGM and LPS on the proliferation of RAW264.7 cells. The endotoxin LPS were selected to stimulate the inflammatory mediator secretions by RAW264.7 macrophages in the present model. Each cell population (2 × 103 cells/ml) was treated with various concentrations of U-OKGM and LPS, respectively. ***p < 0.001, compared with the control group (n = 6).
Figura 5. Efecto de U-OKGM y LPS en la proliferación de células RAW264.7. En este modelo, la endotoxina LPS fue seleccionada para estimular las secreciones mediadoras inflamatorias de los macrófagos RAW264.7. Cada población de células (2 × 103 células/ml) fue tratada con varias concentraciones de U-OKGM y LPS, respectivamente. ***p < 0.001, comparada con el grupo de control (n = 6).
Figure 6. Effect of U-OKGM on macrophage phagocytosis (A), NO production (B), TNF-α production (C) and IL-1β production (D) in RAW264.7 cells with LPS. *p < 0.05, **p < 0.01, ***p < 0.001 compared with the control group (n = 6), #p < 0.05, ##p < 0.01, ###p < 0.001 compared with LPS treatment group (n = 6).
Figura 6. Efecto de la U-OKGM en la fagocitosis de los macrófagos (a), producción de NO (b), producción de TNF-α (c) y producción de IL-1β (d) en células RAW264.7 con LPS. *p < 0.05, **p < 0.01, ***p < 0.001 comparadas con el grupo de control (n = 6), #p < 0.05, ##p < 0.01, ###p < 0.001, comparadas con el grupo de control LPS (n = 6).
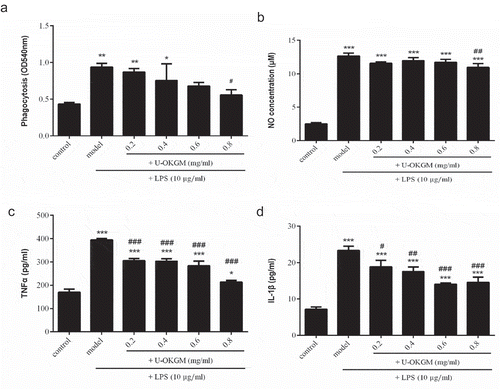
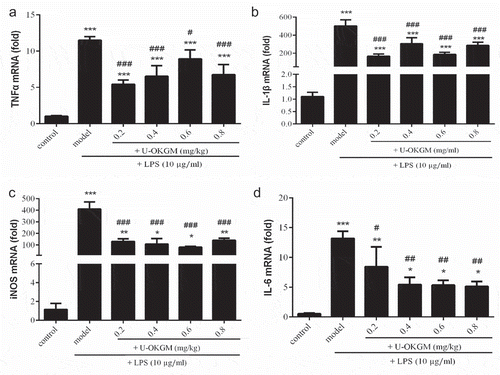
Figure 7. Effect of U-OKGM on TNFα (a), IL-1β (b), iNOS (c) and IL-6 (d) mRNA expression in RAW264.7 cells with LPS. *p < 0.005, **p < 0.01, ***p < 0.001, compared with the control group (n = 3); #p < 0.05, ##p < 0.01, ###p < 0.001 compared with LPS treatment group (n = 3).
Figura 7. Efecto del U-OKGM en TNFα (a), IL-1β (b), iNOS (c) e IL-6 (d) en la expresión del ARNm en células RAW264.7 con LPS. *p < 0.005, **p < 0.01, ***p < 0.001, comparada con el grupo de control (n = 3); #p < 0.05, ##p < 0.01, ###p < 0.001 comparada con el grupo de tratamiento LPS (n = 3).