Abstract
The objective of this work was to develop a polymerase chain reaction (PCR) multiplex technology to detect Salmonella spp. and Escherichia coli O157:H7 in bovine carcasses. To obtain the PCR products, slt-I, slt-II, eaeA and uidA for E. coli and invA for Salmonella spp. genes were amplified in the same reaction. Using this method, microbial presence was investigated in carcasses from Mexican slaughterhouses, and traditional microbiological methods were used to corroborate PCR technology. Sixty samples were collected from municipal slaughterhouses and 60 from federal inspected slaughterhouses (TIF for its initials in Spanish) after sacrifice. Two positive samples (1.6%) were obtained of Salmonella spp. using both techniques in the municipal slaughterhouses. In the samples from both origins, there were positives. The PCR technique proved effective for the rapid and simultaneous identification of microorganisms.
El objetivo de este trabajo fue desarrollar una técnica de reacción en cadena de la polimerasa (PCR) múltiple para la detección simultánea de Salmonella spp. y Escherichia coli O157:H7 en canales de bovino. Para obtener los productos de PCR, se amplifican los genes slt-I, slt-II, eaeA y uidA para E. coli y el gen invA para Salmonella en la misma reacción. Con esta metodología se investigó la presencia de estas bacterias en canales de rastros de México y se corroboraron los resultados con métodos microbiológicos tradicionales. Sesenta muestras de canales de bovino se recogieron en un rastro municipal y 60 en uno Tipo Inspección Federal (TIF) después del sacrificio. De las muestras del rastro municipal se obtuvieron dos positivas (1,6%) a Salmonella spp. usando ambas técnicas. No se encontraron muestras positivas a E. coli O157:H7 por ambas técnicas. La técnica PCR resultó ser rápida y efectiva para la identificación simultánea de microorganismos.
Palabras clave:
Introducción
La presencia de microorganismos patógenos en los alimentos es uno de los problemas importantes en salud pública. Las enfermedades que estos producen son una de las causas más importantes de enfermedades transmitidas por alimentos. Durante la década pasada, los brotes de enfermedades de transmisión alimentaria (ETAs) en humanos estuvieron asociados con el consumo de productos frescos (Bhagwat, Citation2003; Borch & Arinder, Citation2002). Seis microorganismos patógenos están asociados con el 90% de las muertes relacionadas con alimentos en los Estados Unidos de Norteamérica: Salmonella (31%); Listeria (28%); Toxoplasma (21%); Enterovirus (7%); Campylobacter (5%); y Escherichia coli O157:H7 (3%) (White, Zhao, Simjee, Wagner, & McDermott, Citation2002). Salmonella spp.; Staphylococcus aureus; Bacillus cereus; y Clostridium botulinum han sido asociados con enfermedades de origen alimentario por décadas. El serotipo O157:H7 de E. coli ha sido asociado con enfermedades hemorrágicas desde los inicios de los años ochenta y ha causado gran cantidad de brotes de ETAs en Norteamérica, Europa y Japón (White et al., Citation2002), siendo los rumiantes el reservorio de este patógeno. Las rutas de contaminación han sido de persona a persona; por alimentos; contacto con el medioambiente de los ranchos o incluso por agua contaminada (Borch & Arinder, Citation2002). Los programas de aseguramiento de la calidad que implementan controles microbiológicos son muy beneficiosos pues ayudan a reducir el riesgo de infección de los consumidores. Los métodos microbiológicos para patógenos, conforme a las Normas Oficiales Mexicanas y los métodos convencionales, se basan en pruebas estandarizadas para su aislamiento e identificación y requieren normalmente el uso de medios de pre-enriquecimientos no selectivos; enriquecimientos selectivos; aislamiento en medios selectivos; y posteriormente la confirmación con pruebas bioquímicas y serológicas específicas para cada microorganismo, por lo que estos métodos son muy laboriosos y requieren de más tiempo para obtener los resultados.
Como una posible solución a estos inconvenientes, han surgido de la investigación varias metodologías, entre las que resaltan los métodos inmunológicos y moleculares. En los últimos años, métodos basados en la técnica de reacción en cadena de la polimerasa (PCR) se han convertido en la herramienta diagnóstica más popular en microbiología (Olsen, Citation2000) debido a su especificidad, sensibilidad, rapidez, detección y selectividad. La propuesta de este estudio fue desarrollar una metodología de amplificación de ADN múltiple por PCR para identificar la presencia de genes de patogenicidad y virulencia de Escherichia coli enterohemorrágica O157:H7 y Salmonella spp. en canales de res, y cuantificar la presencia de estas bacterias en rastros de México, comparando los resultados con métodos microbiológicos oficiales.
Materiales y métodos
Muestreo
Se realizó un muestreo aleatorio simple de la región del pecho de 120 canales de res con un hisopo estéril: 60 canales en un Rastro Municipal (Tlalnepantla, Estado de México), y 60 canales en un rastro Tipo Inspección Federal (TIF) número 45, Empacadora de Carnes Unidad Ganadera de Aguascalientes, UGASA (Aguascalientes, México).
El número de muestras se determinó de acuerdo con los criterios microbiológicos para carne cruda establecidos por la International Commission on Microbiological Specifications for Foods (ICMSF, Citation1984). Las categorías de los planes de muestreo se determinaron con base en el riesgo para la salud humana que representan la Salmonella spp. y E. coli O157:H7, así como en las condiciones previsibles de manejo y consumo a las que estará sujeta la carne de res tras el muestreo, las cuales modificarán el riesgo; en este caso, la condición que reducirá el riesgo bacteriológico en la carne de res es la cocción.
La toma de muestra de los canales se realizó al finalizar el faenado de los animales, usando la metodología propuesta por Bhaduri y Cottrell (Citation1998). Se identificó el codo y se trazó una línea imaginaria hacia el dorso, pasando por la línea media del codo, representando el punto de inicio. Un molde de 300 cm3 se colocó en esta área para tomar cada muestra, se frotó el hisopo estéril en las superficies que comprendía el molde, en seguida los hisopos se colocaron en tubos de polipropileno con medio de transporte estéril y fueron trasladados del rastro al laboratorio para su procesamiento. Las muestras se transportaron de acuerdo al Manual de prácticas. Curso: Toma y manejo de muestras para análisis bacteriológico (Laboratorio Nacional de Salud Pública, Citation1992).
Técnica de PCR
Obtención de ADN bacteriano
Para obtener ADN de las bacterias se utilizó el método de Silhavy, Berman y Enquist (Citation1984). Se utilizaron como cepas de referencia para Salmonella: S. typhi JM1301; S. typhimurium F1; S. typhimurium F2; S. typhimurium ATCC; S. gallinarum FVB323; y S. enteritidis F4. Para Escherichia coli O157:H7: 91984, 93206 y 93211, donadas por la Facultad de Medicina, UNAM. La prueba de especificidad para la reacción de PCR múltiple se realizó mediante la utilización de ADN de otros géneros bacterianos relacionados con los estudiados: Shigella sonnei; Proteus sp.; Klebsiella sp.; Escherichia coli: 55784; 66295; 88/66; 88265; E2348/69; y JPN10. Para la determinación de la sensibilidad de la técnica de PCR se utilizaron cultivos a partir de cepas certificadas de Escherichia coli O157:H7 y Salmonella spp. A partir del ADN cuantificado por flurometría se efectuaron diluciones décuples y dobles hasta obtener 0,0000468 nanogramos (ng) de DNA/μl que equivalen a 0,312 unidades formadoras de colonia (UFC), tomando en cuenta que una bacteria contiene aproximadamente 3×106 pares de bases (pb) de DNA (Bhaduri & Cottrell, Citation1998).
Amplificación múltiple por PCR para la detección simultánea de secuencias diagnósticas de Salmonella spp. y Escherichia coli O157:H7
Se realizó amplificación múltiple de los genes que codifican para las toxinas Shiga-like (slt-I y slt-II), el gen eaeA y el gen uidA de E. coli O157:H7 (Cebula, Payne, & Feng, Citation1995; Meng, Zhao, Doyle, Mitchell, & Kresovich, Citation1997). En el caso de Salmonella spp. se utilizó el gen de invasión A (invA) (Rahn et al., Citation1992). Además, se emplearon un par de iniciadores para identificar al plásmido Bluscript (pBS) como control positivo. Las secuencias de los iniciadores empleados se presentan en el .
Tabla 1. Iniciadores empleados.
. Oligonucleotide used.
Las condiciones de amplificación fueron en un inicio las referidas por los autores antes mencionados, para cada uno de los dos microorganismos y el DNA del plásmido, posteriormente estas condiciones se adecuaron realizando PCR múltiple para la identificación simultánea (Cebula et al., Citation1995; Meng et al., Citation1997; Rahn et al., Citation1992; Gooding et al., Citation1999).
En la reacción en cadena de la polimerasa se emplearon 20 μl la mezcla de reacción; se utilizó 0,0000075 ng de ADN/μl; el amortiguador de reacción a una concentración final de 1,5 mM; una mezcla de cada uno de los cuatro deoxinucleótidos a una concentración final de 0,2 mM, 1 μM de cada uno de los iniciadores y 0,0625 U/μl de la enzima Taq polimerasa. La reacción se realizó en 35 ciclos en un termociclador a 94°C por 30 s; 50°C por 30 s; y 72°C por 30 s. Los productos de amplificación fueron analizados en geles de agarosa al 3% como concentración final y teñidos con bromuro de etidio.
En el laboratorio, con ocho muestras de carne de res se realizaron contaminaciones con Escherichia coli O157:H7 y Salmonella spp. cada una por separado tanto con carne lavada con solution de buffer de fosfatos (PBS), como con carne sin lavar, conformando las cuatro primeras muestras; la quinta y sexta muestras incluían a ambos microorganismos, con carne limpiada con PBS y sin limpiar. Además se incluyeron dos muestras más, una de las cuales se limpió con PBS y la otra no, sin la contaminación con los microorganismos antes mencionados. De estos grupos experimentales se tomaron muestras para realizar PCR para la identificación de los microorganismos en estudio.
Identificación microbiológica oficial
La detección microbiológica de Salmonella spp., se realizó conforme lo señala la Norma Oficial Mexicana: Método para la determinación de Salmonella en alimentos (NOM-114- SSA1-1994); y la determinación de Escherichia coli O157:H7 se realizó de acuerdo a la metodología validada por la Association of Official Analitical Chemists (Helrich, Citation1990).
Resultados
Aislamientos de Salmonella spp. y E. coli 0157:H7
Empleando métodos microbiológicos en el rastro TIF no se aisló ninguna colonia bacteriana sospechosa. En cambio, en el rastro municipal se obtuvo una muestra positiva a Salmonella spp. y ninguna a E. coli O157:H7. En las , y se muestran los resultados de las pruebas microbiológicas realizadas, indicando el número de colonias sospechosas a Salmonella spp. y los resultados obtenidos de las pruebas bioquímicas y serológicas.
Tabla 2. Colonias sospechosas de Salmonella spp. aisladas en medios selectivos provenientes de muestras del rastro municipal.
. Colonies suspected of having Salmonella spp. isolated in selective cultures of meat samples from Mexican municipal slaughterhouses.
Tabla 3. Identificación presuntiva por pruebas bioquímicas de colonias sospechosas de Salmonella spp. provenientes de muestras del rastro municipal.
. Identification by biochemical test from colonies suspected of having Salmonella spp. from meat samples from Mexican municipal slaughterhouses.
Tabla 4. Prueba de aglutinación con suero polivalente para Salmonella en colonias sospechosas en muestras provenientes del rastro municipal.
. Agglutination test with polyvalent serum for Salmonella in colonies suspected from having it, in meat samples from Mexican municipal slaughterhouses.
Amplificación múltiple por PCR para la detección simultánea de secuencias diagnósticas de Salmonella spp. y Escherichia coli O157:H7
Los resultados obtenidos de PCR con las muestras de carne contaminadas experimentalmente, fueron la amplificación de todos los genes en estudio.
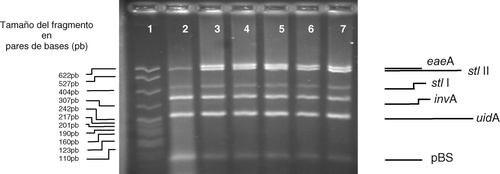
En la se muestra el resultado de la amplificación simultánea de los genes de Escherichia coli O157:H7 y Salmonella empleando diferentes concentraciones de ADN de ambos microorganismos (desde 15 hasta 0,00015 ng de ADN).
Figura 1. Amplificación múltiple por PCR para Escherichia coli O157:H7 y Salmonella spp. con diferentes concentraciones de DNA. Los genes amplificados son eaeA, uidA, stlI, stlII, invA y pBS, correspondientes a E. coli (los cuatro primeros), Salmonella spp. y pBS respectivamente. Gel de agarosa al 3% teñido con bromuro de etidio 0,5μg/ml. PCR con dos mM de Mg++. En el carril uno se encuentra el marcador de peso molecular correspondiente a pBR322/Msp1. En el carril dos se observa la amplificación múltiple con 0,00000015 ng de DNA/μl; en el carril tres se observa la amplificación múltiple con 0,00015 ng de DNA/μl; en el carril cuatro se observa la amplificación múltiple con 0,0015 ng de DNA/μl; en el carril cinco se observa la amplificación múltiple con 0,15 ng de DNA/μl; en el carril seis se observa la amplificación múltiple con 15 ng de DNA/μl. En el carril siete, control positivo.
Figure 1. Multiple PCR amplification for E. coli O157:H7 y Salmonella spp. using different concentrations of DNA. The genes to amplify are eaeA, uidA, stlI, stlII, invA y pBS, from E. coli (the first four) and Salmonella spp. and pBS respectively. Agarose gel at 3% tainted with ethidium bromide at 0.5 mg/ml. PCR with two mM of Mg++. Track one: molecular weight marker pBR322/Msp1. Track two: multiple amplification 0.00000015 ng de ADN/ml. Track three: multiple amplification 0.00015 ng de ADN/ml. Track four: multiple amplification 0.0015 ng de ADN/ml. Track five: multiple amplification 0.15 ng of ADN/ml. Track six: multiple amplification 15 ng de ADN/ml. Track seven: positive control.
Discusión
Las carnes crudas contaminadas son uno de los principales vehículos de transmisión de enfermedades bacterianas, ya que Escherichia coli O157:H7 y Salmonella spp. han estado implicadas en una gran cantidad de brotes; Salmonella spp. con un estimado de 1,3 billones de casos alrededor del mundo (Pang, Bhutta, Finlay, & Altwegg, Citation1995) y la hemorrágica Escherichia coli con la cepa O157:H7 es el serotipo más frecuente implicado en las enfermedades bacterianas en el mundo (Feng & Monday, Citation2000). En este estudio se presenta una amplificación múltiple por PCR para la detección de estos dos patógenos muy comunes en las infecciones y toxi-infecciones por consumo de carne.
Existen estudios previos que han usado PCR múltiple o PCR de tiempo real para detectar Salmonella y E. coli O157:H7 en carne (Sharma & Carlson, Citation2000) o Salmonella spp. y Listeria monocytogenes en carnes procesadas con un enriquecimiento previo de 6 a 18 horas (Wang, Jothikumar, & Griffiths, Citation2004). Vollenhofer-Schrumpf et al. (Citation2005) encontraron los mismos resultados cuando compararon métodos microbiológicos tradicionales con los obtenidos por PCR, detectando 1–10 UFC/25g de cada patógeno añadido, encontrando cuatro patógenos simultáneamente. La detección rápida, oportuna y confiable de los diversos patógenos bacterianos en cualquier etapa de la cadena cárnica, constituye un paso importante para su control, vigilancia, monitoreo y prevención. Los iniciadores para la identificación específica de E. coli O157:H7, identificando simultáneamente los genes que codifican para las toxinas que produce son Shiga-like (slt-I y slt-II) y el gen eaeA que codifica la producción de una proteína que permite a la bacteria adherirse estrechamente a la célula, llamada intimina; además de la amplificación del gen uidA el cual codifica para la β-glucoronidasa (Cebula et al., Citation1995; Meng et al., Citation1997). Es indispensable la amplificación de estos cuatro iniciadores para identificar el serotipo O157:H7. En el caso de Salmonella spp. se necesita el gen de invasión A (invA) que es parte de un grupo de genes que le permiten penetrar a las células hospedadoras (Rahn et al., Citation1992).
En el presente estudio, la técnica de PCR mostró ser efectiva (detectó dos canales positivos a Salmonella spp.) en comparación con el método microbiológico oficial, con el cual sólo una de ellas fue confirmada. Cabe hacer mención que la técnica de PCR no es indicativa de la viabilidad de los microorganismos que puedan estar presentes en los alimentos. Por otro lado, ni con la técnica de PCR ni con el método validado por la AOAC se pudieron encontrar muestras positivas a Escherichia coli O157:H7. En México se ha emitido gran cantidad de documentos normativos que permiten que en los procesos que llevan a cabo las industrias de alimentos, se observen y mantengan lineamientos a cumplir desde el punto de vista sanitario, como es el caso de la emisión de la Norma Oficial Mexicana NOM-114-SSA1-1994.
Con respecto a la sensibilidad, en el presente trabajo se utilizó para realizar PCR múltiple, 0,0000075 ng de ADN/μl ó 5 UFC. Meng et al. (Citation1997); Venkateswaran, Kamijoh, Ohashi y Nakanishi (Citation1997); y Cebula et al. (Citation1995) no reportan sensibilidad para sus trabajos con E. coli O157:H7. Sin embargo, Uyttendale, van Boxsttael y Debevere (Citation1999) señala una sensibilidad para la misma bacteria de 0,001 ng de ADN/μl ó 103 UFC por reacción de PCR. Para el gen de invasión en Salmonella spp., Rahn et al. (Citation1992) reportan una sensibilidad de 0,0027 ng de ADN/μl, equivalente a 180 UFC.
En el actual comercio nacional e internacional se requieren métodos y técnicas efectivas que permitan la detección de microorganismos de manera segura, siendo indispensable que cuando se importe carne o animales en pie, se pueda verificar que estén libres de patógenos y/o de sus toxinas en el mismo lugar de recepción como puertos, aeropuertos, fronteras o lugares de entrada en general, donde se tengan los cuidados y precauciones necesarios para poder realizar las determinaciones microbiológicas. Estos lugares no tienen la capacidad de almacenar la carne por largo tiempo para la posterior realización de las pruebas microbiológicas convencionales, por lo cual podrían estar introduciendo al país carne sin esta evaluación. El realizar este tipo de prueba, garantizaría el control y agilizaría la movilización de los animales o sus productos. Al obtener resultados positivos a partir de las muestras de alimentos, sólo se requeriría realizar los métodos confirmativos a un menor número de muestras, lo que se traduciría en un menor gasto.
Conclusiones
En este estudio se realizó amplificación múltiple por PCR para la detección de patógenos en alimentos (E. coli 0157:H7 y Salmonella spp.), mostrando ser una técnica rápida y efectiva de identificación. La eficiencia demostrada por la PCR ha sido mejor al compararse con los métodos microbiológicos de referencia. Por tanto, esta metodología puede constituir una estrategia útil para la detección rápida y sensible de patógenos en alimentos y puede emplearse como una herramienta adicional al rango de herramientas diagnósticas disponibles para el estudio de patógenos de origen alimentario.
Agradecimientos
El presente estudio estuvo financiado por el Concejo Nacional de Ciencia y Tecnología (CONACyT) mediante el proyecto 27706B.
Referencias
- Bhaduri , S. and Cottrell , B. 1998 . A simple preparation method from various foods for PCR detection of pathogenic. Yersinia enterocolitica: a possible model for other food pathogens . Molecular and Cellular Probes , 12 : 79 – 83 .
- Bhagwat , A. A. 2003 . Simultaneous detection of Escherichia coli O157:H7, Listeria monocytogenes and Salmonellastrains by real-time PCR . International Journal of Food Microbiology , 84 : 217 – 224 .
- Borch , E. and Arinder , P. 2002 . Bacteriological safety issues in red meat and ready-to-eat meat products, as well as control measures . Meat Science , 62 : 381 – 390 .
- Cebula , T. A. , Payne , W. L. and Feng , P. 1995 . Simultaneous identification of strains of E. coli serotype O157:H7 and their shiga-like toxin type by mismatch amplification mutation assay – multiple PCR . Journal of Clinical Microbiology , 31 ( 1 ) : 248 – 250 .
- Feng , P. and Monday , S. R. 2000 . Multiple PCR for detection of trait and virulence factors in enterohemorrhagic Escherichia coli serotypes . Molecular and Cellular Probes , 14 : 333 – 337 .
- Gooding , C. M. and Choudary , P. V. 1999 . Comparison of different primers for rapid detection of Salmonella using the PCR . Molecular and Cellular Probes , 13 : 341 – 347 .
- Helrich K. Official methods of analysis of the Association of Official Analytical Chemists , 15ava ed. 1990 Bacteriological Analitic. Arlington, USA
- ICMSF. 1984 . Microorganismos de los alimentos (Vol. 2). Métodos de muestreo para análisis microbiológicos: Principios y aplicaciones específicas , Zaragoza, , España : Acribia .
- Laboratorio Nacional de Salud Pública. 1992 . Manual de prácticas. Curso: Toma y manejo de muestras para análisis bacteriológico , México, DF : Subsecretaría de Regulación y Fomento Sanitario, Secretaría de Salud Pública .
- Meng , J. , Zhao , S. , Doyle , M. P. , Mitchell , S. E. and Kresovich , S. 1997 . A multiple PCR for identifying shiga-like toxin producing E. coli O157:H7 . Letters in Applied Microbiology , 24 : 172 – 176 .
- Olsen , E. J. 2000 . DNA-based methods for detection of food-borne bacterial pathogens . Food Research International , 33 : 257 – 266 .
- Pang , T. , Bhutta , Z. A. , Finlay , B. B. and Altwegg , M. 1995 . Typhoid fever and other salmonellosis: A continuing challenge . Trends in Microbiology , 3 : 253 – 255 .
- Rahn , K. , De Grandis , S. A. , Clarke , R. C. , McEwen , S. A. , Galán , J. E. , Ginocchio , C. , Curtiss III , R. and Gyles , C. L. 1992 . Amplification of invA gene sequence of Salmonella typhimurium by PCR as a specific method of detection of Salmonella . Molecular and Cellular Probes , 6 : 271 – 279 .
- Secretaría de Salud . 1994 . Bienes y servicios. Método para la determinación de Salmonella en alimentos 2001–2003 (NOM-114-SSA1-1994) 22 de septiembre de 1995
- Sharma , V. K. and Carlson , S. A. 2000 . Simultaneous detection of Salmonella strains and Escherichia coli O157:H7 with fluorogenic PCR and single-enrichment-broth culture . Applied and Environmental Microbiology , 66 ( 12 ) : 5472 – 5476 .
- Silhavy , T. J. , Berman , M. L. and Enquist , L. W. 1984 . “ Experiments with gene fusions ” . Cold Spring Harbor Laboratory, Cold Spring Harbor, N.Y
- Uyttendale , M. , van Boxsttael , S. and Debevere , J. 1999 . PCR assay for detection of the E. coli O157:H7 eae gene and effect of the sample preparation method on PCR detection of heat-killed E. coli O157:H7 in ground beef . International Journal of Food Microbiology , 52 : 85 – 95 .
- Venkateswaran , K. , Kamijoh , Y. , Ohashi , E. and Nakanishi , H. 1997 . A simple filtration technique to detect enterohemorragic E. coli O157:H7 and its toxin in beef by multiplex PCR . Applied and Environmental Microbiology , 63 ( 10 ) : 4127 – 4131 .
- Vollenhofer-Schrumpf , S. , Buresch , R. , Unger , G. , Stahl , N. , Fränzl , G. and Schinkinger , M. 2005 . Detection of Salmonella spp., Escherichia coli O157:H7, Listeria monocytogenes and Campylobacter spp. in chicken samples by multiple polymerase chain reaction and hybridization using the genegen major food pathogens detection kit . Journal of Rapid Methods and Automation in Microbiology , 13 ( 3 ) : 148 – 176 .
- Wang , X. W. , Jothikumar , N. and Griffiths , M. W. 2004 . Enrichment and DNA extraction protocols for the simultaneous detection of Salmonella and Listeria monocytogenes in raw sausage meat with multiplex real-time PCR . Journal of Food Protection , 67 ( 1 ) : 189 – 192 .
- White , D. G. , Zhao , S. , Simjee , S. , Wagner , D. D. and McDermott , F. P. 2002 . Antimicrobial resistance of foodborne pathogens . Microbes and Infection , 4 : 405 – 412 .