Figures & data
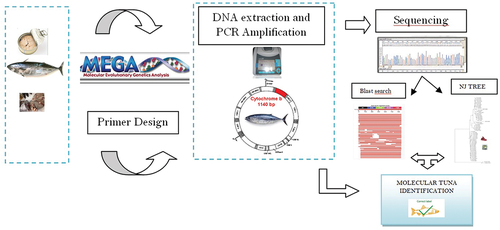
Table 1. Authenticity determination of Tuna species in commercial fresh and canned product.
Tabla 1. Determinación de la autenticidad de las especies de atún en productos comerciales frescos y en conserva
Table 2. Comparison of DNA yield, purity, PCR positive amplification for several foodstuffs of Tuna extracted with seven different methods studied.
Tabla 2. Comparación del rendimiento del ADN, la pureza y la amplificación positiva de la RCP para varios productos alimenticios de atún extraídos con siete métodos diferentes estudiados
Figure 1. Example of Electrophoretic analysis gel (1% agarose) of PCR products of the cytochrome b fragments from left to right. Lanes 1 and 8: Amplification of DNA of canned tuna fish, Mq: Lanes 9 molecular weight marker 100-bp ladder; PCR products for tuna fresh samples: sample reference lanes 10: Katsowonus pelamis, lanes 11–12 Thunnus Thynnus, lanes 13: Thunnus albacares, lanes 14: Euthynnus alleteratus, lanes 15: Auxis rochei and lanes 16: Sarda sarda.
Figura 1. Ejemplo de gel de análisis electroforético (agarosa al 1%) de los productos de RCP en los fragmentos del citocromo b, de izquierda a derecha. Filas 1 y 8: amplificación del ADN del atún en conserva; Mq: filas 9 marcador de peso molecular, escalera de 100-bp; productos RCP para muestras de atún fresco: filas de referencia de la muestra 10: Katsowonus pelamis; filas 11–12 Thunnus Thynnus; filas 13: Thunnus albacares; filas 14: Euthynnus alleteratus; filas 15: Auxis rochei; y filas 16: Sarda sarda.
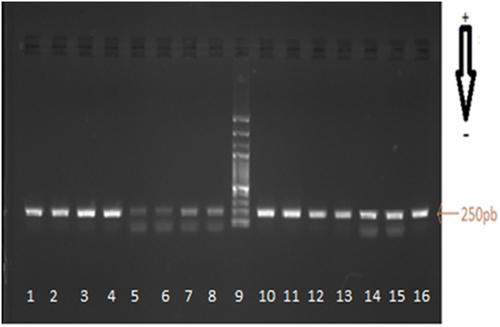
Figure 2. Phylogenetic tree obtained from sequence divergence of a Cytb gene for 53 sequences of Tuna. NJ tree inferred from TN distances between sequences of partial Cytb gene in 25 samples of Katsowonus pelamis Sequences starting with “TKP” species, 10 samples of Thunnus Thynnus Sequences starting with “TR” species, 8 samples Thunnus albacares sequences starting with “TYF” species, 5 samples Euthynnus alleteratus sequences starting with “TEU” species, 1 sample Auxis rochei sequences starting with “TAX” species, 1 sample Sarda sarda Sequences starting with “TSD” species and the sequenced gene referenced in the NCBI GenBank; Numbers denote bootstrap percentages based on 1500 replications from neighbor-joining analysis.
Figura 2. Árbol filogenético obtenido a partir de la divergencia de secuencias de un gen Cytb para 53 secuencias de atún. Árbol NJ inferido a partir de las distancias TN entre las secuencias del gen Cytb parcial en 25 muestras de Katsowonus pelamis; secuencias que comienzan con la especie “TKP”, 10 muestras de Thunnus Thynnus; secuencias que comienzan con la especie “TR”, ocho muestras de Thunnus albacares; secuencias que comienzan con la especie “TYF”, cinco muestras de Euthynnus alleteratus; secuencias que comienzan con la especie “TEU”, una muestra de Auxis rochei; secuencias que comienzan con la especie “TAX”, una muestra de Sarda sarda; secuencias que comienzan con la especie “TSD” y el gen secuenciado referenciado en el NCBI GenBank. Los números denotan los porcentajes de bootstrap basados en 1,500 réplicas del análisis de unión de vecinos.
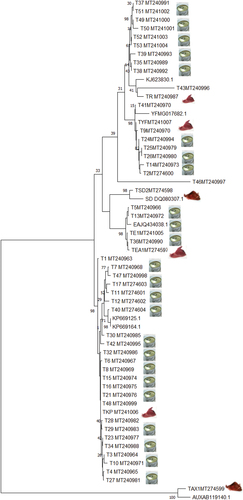
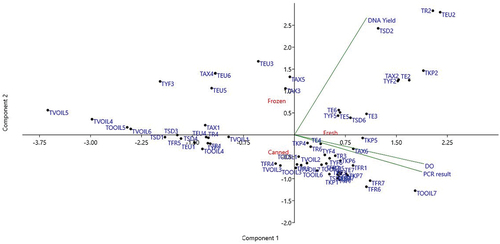
Figure 3. Loading plots from partial least square PLS regression carried out on all X–variables DO: A260/A280, DNA YIELD, EX1: Kit fast DNA method, EX2: PCI/SDS method, EX3: SDS/PCI/NaCl method, EX4: CHELEX method, EX5: CTAB/CI method, EX6: TRITON/CI method, EX7: DNeasy Tissue kit method and all tuna foodstuff and the Y-variable (PCR RESULT: PCR amplification). All data in the PCA projections represent the means value of 6 replicates for each parameter.
Figura 3. Gráficos de carga de la regresión de mínimos cuadrados parciales realizada en todas las variables X DO: A260/A280, rendimiento de ADN, EX1: Método de ADN rápido del kit, EX2: Método PCI/SDS, EX3: Método SDS/PCI/NaCl, EX4: Método CHELEX, EX5: Método CTAB/CI, EX6: Método TRITON/CI, EX7: Método del kit DNeasy Tissue y todos los alimentos del atún y la variable Y (resultado de la RCP: amplificación de la RCP). Todos los datos de las proyecciones del PCA representan el valor medio de seis réplicas para cada parámetro.
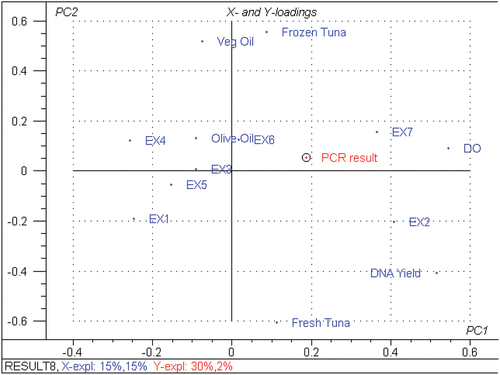
Figure 4. Loading plots from The Principal Component Analysis (PCA) carried out on all variables DO: A260/A280, DNA YIELD, and PCR RESULT: PCR amplification). All data in the PCA projections represent the means value of 6 replicates for each parameter.
Figura 4. Gráficos de carga del análisis de componentes principales (ACP) realizado sobre todas las variables DO: A260/A280, DNA YIELD, y RCP resultado: amplificación RCP. Todos los datos de las proyecciones del PCA representan el valor medio de seis réplicas para cada parámetro.